Künstliche Intelligenz (KI) hilft dabei, den Stammbaum von Viren neu zu zeichnen. Vorhergesagte Proteinstrukturen, die mit AlphaFold und chatbot-inspirierten „Protein-Sprachmodellen“ erstellt wurden, haben überraschende Verbindungen in einer Familie von Viren aufgedeckt, die Pathogen umfasst, die Menschen infizieren sowie aufkommende Bedrohungen.
Ein großer Teil des Verständnisses der Wissenschaftler über virale Evolution basiert auf dem Vergleich von Genomen. Allerdings zeigt die blitzschnelle Evolution von Viren, insbesondere von solchen mit RNA-Genomen, und deren Neigung, genetisches Material von anderen Organismen zu übernehmen, dass genetische Sequenzen tiefere und entfernte Beziehungen zwischen Viren verbergen können, die je nach untersucht gen wird, variieren können.
Im Gegensatz dazu ändern sich die Formen oder Strukturen der durch virale Gene kodierten Proteine tendenziell langsam, was es ermöglicht, diese versteckten evolutionären Verbindungen zu erkennen. Bis zum Aufkommen von Werkzeugen wie AlphaFold, das Proteinstrukturen in großem Maßstab vorhersagen kann, war es jedoch nicht möglich, Proteinstrukturen über eine gesamte Virusfamilie hinweg zu vergleichen, sagt Joe Grove, ein Molekularvirologe an der Universität Glasgow, Großbritannien.
In einem diese Woche veröffentlichten Artikel in Nature1 zeigen Grove und sein Team die Kraft eines strukturbasierten Ansatzes bei den Flaviviren — einer Gruppe, die das Hepatitis-C-, Dengue- und Zika-Virus sowie einige bedeutende Tierpathogene und Arten umfasst, die aufkommende Bedrohungen für die menschliche Gesundheit darstellen könnten.
Wie Viren eindringen
Das Verständnis der Forscher über die evolution der Flaviviren basiert hauptsächlich auf Sequenzen langsam evolvierender Enzyme, die ihr genetisches Material kopieren. Es ist jedoch bemerkenswert wenig bekannt über die Ursprünge der „viral entry“-Proteine, die Flaviviren zum Eindringen in Zellen verwenden und die den Wirt bestimmen, den sie infizieren können. Grove argumentiert, dass diese Wissenslücke die Entwicklung eines wirksamen Impfstoffs gegen Hepatitis C, der jährlich Hunderttausende von Menschen tötet, verlangsamt hat.
„Auf der Sequenzebene sind die Dinge so divergiert, dass wir nicht sagen können, ob sie verwandt sind oder nicht“, sagt er. „Der Durchbruch in der Vorhersage von Proteinstrukturen öffnet die gesamte Frage, und wir können die Dinge ziemlich klar sehen.“
Die Forscher verwendeten DeepMinds AlphaFold2-Modell und ESMFold, ein Struktur-Vorhersagetool, das von dem Technologiegiganten Meta entwickelt wurde, um mehr als 33.000 vorhergesagte Strukturen für Proteine von 458 Flavivirus-Spezies zu generieren. ESMFold basiert auf einem Sprachmodell, das mit zig Millionen von Proteinsequenzen trainiert wurde. Im Gegensatz zu AlphaFold benötigt es nur eine Eingabesequenz, anstatt sich auf mehrere Sequenzen ähnlicher Proteine zu stützen, was es besonders nützlich für die Untersuchung der mysteriösesten Viren machen könnte.
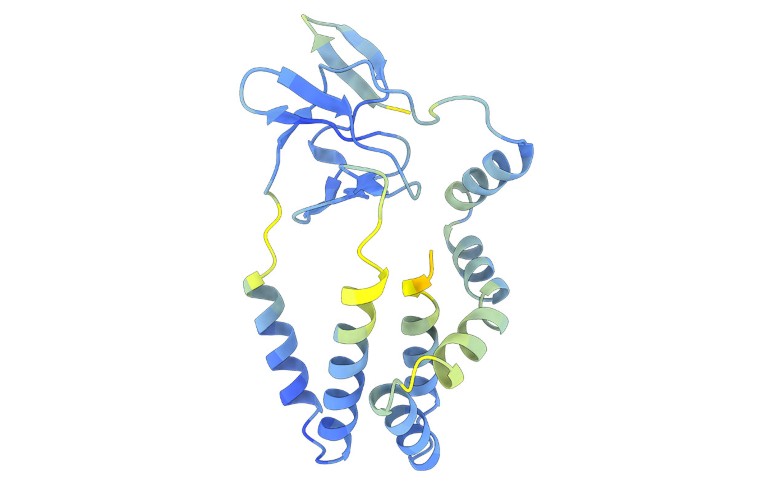
Die vorhergesagten Strukturen ermöglichten es den Autoren, virale Entry-Proteine zu identifizieren, deren Sequenzen sich stark von denen bekannter Flaviviren unterscheiden. Sie fanden einige unerwartete Verbindungen. So verwendet die Virusgruppe, die Hepatitis C umfasst, ein System, um Zellen zu infizieren, das dem ähnelt, das sie bei den Pestiviren entdeckt haben — einer Gruppe, die das klassische Schweinegrippevirus umfasst, das hämorrhagisches Fieber bei Schweinen verursacht, und andere Tierpathogene.
Die KI-gestützten Vergleiche zeigten, dass dieses Eingabesystem sich von dem vieler anderer Flaviviren unterscheidet. „Für Hepatitis C und seine Verwandten wissen wir nicht, woher ihr Eingabesystem kommt. Es könnte erfunden worden sein“, sagt Grove.
Von Bakterien gestohlen
Die vorhergesagten Strukturen zeigten auch, dass die gut untersuchten Eingangsproteine von Zika- und Dengue-Viren die gleichen Ursprünge haben wie die der „seltsamen und wunderbaren“ Flaviviren mit riesigen Genomen, einschließlich des Haseki-Zeckenvirus, das Fieber bei Menschen auslösen kann. Eine weitere große Überraschung war die Entdeckung, dass einige Flaviviren ein Enzym besitzen, das anscheinend von Bakterien gestohlen wurde.
„Das wäre beispiellos“, sagt die Virologin Mary Petrone von der Universität Sydney, Australien, wäre es nicht für die Entdeckung ihres Teams in diesem Jahr eines ähnlichen Diebstahls bei einer besonders „seltsamen und wunderbaren“ Flavivirus-Art2. „Genetischer Piraterie könnte eine größere Rolle bei der Evolution der Flaviviren gespielt haben, als bisher gedacht“, fügt sie hinzu.
David Moi, ein computergestützter Biologe an der Universität Lausanne, Schweiz, sagt, dass die Flavivirus-Studie lediglich die Spitze des Eisbergs ist und dass die evolutionären Geschichten anderer Viren und sogar einiger zellulärer Organismen wahrscheinlich mit KI neu erzählt werden. „Jetzt, da wir einen Blick weiter werfen können, müssen all diese Dinge ein kleines Update bekommen“, sagt er.

 Suche
Suche
 Mein Konto
Mein Konto

